Tutorial
Gonzalo E. Pinilla-Buitrago
2025-06-17
Source:vignettes/tutorial-1-intro-es.Rmd
tutorial-1-intro-es.RmdPaquete fastbioclim: Derivación eficiente de
variables bioclimáticas
El paquete fastbioclim está diseñado para generar
variables bioclimáticas de manera eficiente, utilizando dos flujos de
trabajo (workflows) distintos.
En Memoria (“terra”): El primer método se basa en el
paquete terra y es ideal cuando los rásters pueden ser
procesados completamente en la memoria RAM del computador.
Fuera de Memoria (“tiled”): El segundo método está
diseñado para grandes rásters. Divide el área de interés en mosaicos (o
cuadrículas) que son procesados de forma independiente utilizando los
paquetes exactextractr y Rfast. Este enfoque
out-of-core (fuera de memoria) permite analizar datos de
cualquier tamaño, sin importar la memoria RAM disponible.
La principal ventaja de fastbioclim es que puede
seleccionar de forma inteligente el método más adecuado con el argumento
method = "auto", garantizando siempre el mejor balance
entre velocidad y uso de memoria.
Además de su rendimiento, fastbioclim ofrece una gran
flexibilidad:
- Permite calcular un subconjunto de variables sin necesidad de generar el conjunto completo.
- Expande el conjunto a 35 variables bioclimáticas, incluyendo radiación solar (bios 20-27) y resúmenes de humedad (bios 28-35) basados en la nomenclatura de ANUCLIM 6.1 (Xu & Hutchinson, 2012).
- Ofrece la opción de definir períodos de tiempo personalizados (ej. semanas, semestres) para las variables basadas en períodos (como bio08 o bio18).
- Permite utilizar un ráster de temperatura promedio real (parámetro
tavg), en lugar de la aproximación estándar de (tmax + tmin) / 2. - Permite analizar cualquier variable temporal (velocidad del viento,
humedad, etc.) con la misma arquitectura potente y escalable, usando la
función
derive_statistics(). - Permite el uso de índices estáticos para un control avanzado, ideal para análisis de series temporales (p. ej., asegurar que el “período más cálido” se refiera siempre a los mismos meses cada año).
La funcionalidad de fastbioclim está inspirada en la
función biovars() del paquete dismo, con el
objetivo de agilizar y escalar el proceso de creación de variables
bioclimáticas para el modelado ecológico y ambiental.
Aviso: Este paquete está en desarrollo
Este paquete de R está actualmente en desarrollo y puede contener errores, fallos o características incompletas.
Se agradecen las contribuciones y los informes de errores. Si encuentra problemas o tiene sugerencias de mejora, por favor abra un issue en el repositorio de GitHub.
Instalación
Para instalar fastbioclim, puede utilizar el paquete
remotes. Si aún no lo tiene instalado, puede hacerlo
ejecutando:
install.packages("remotes")
remotes::install_github("gepinillab/fastbioclim")
# Instalar para obtener los datos de ejemplo
remotes::install_github("gepinillab/egdata.fastbioclim")Instale y cargue los paquetes necesarios:
# Cargue librerías e instálelas si es necesario
if (!require("terra")) {
install.packages("terra")
}
if (!require("future.apply")) {
install.packages("future.apply")
}
if (!require("progressr")) {
install.packages("progressr")
}
if (!require("fastbioclim")) {
remotes::install_github("gepinillab/fastbioclim")
}
if (!require("egdata.fastbioclim")) {
remotes::install_github("gepinillab/egdata.fastbioclim")
}Obtener las 19 variables bioclimáticas para Ecuador
Al igual que biovars(), este paquete requiere que el
usuario provea las variables climáticas promedio por unidad de tiempo
para el cálculo de variables. Tradicionalmente dichas unidades de tiempo
corresponden a promedios mensuales de temperatura y precipitación para
decenas de años. Para este ejemplo vamos a usar variables obtenidas y
procesadas de CHELSA v2.1 (Karger et al., 2017) para Ecuador que están
disponibles dentro del paquete de datos (egdata.fastbioclim
solo en GitHub).
# Obtener lista de rasters y crear un spatRaster para cada variable
# Temperatura mínima
tmin_ecu <- system.file("extdata/ecuador/", package = "egdata.fastbioclim") |>
list.files("tmin", full.names = TRUE) |> rast()
# Temperatura máxima
tmax_ecu <- system.file("extdata/ecuador/", package = "egdata.fastbioclim") |>
list.files("tmax", full.names = TRUE) |> rast()
# Precipitación
prcp_ecu <- system.file("extdata/ecuador/", package = "egdata.fastbioclim") |>
list.files("prcp", full.names = TRUE) |> rast()
# Definir directorio donde se guardarán los rasters
output_dir_bioclim <- file.path(tempdir(), "bioclim_ecuador")
# Obtener las 19 variables para Ecuador
bioclim_ecu <- derive_bioclim(
bios = 1:19,
tmin = tmin_ecu,
tmax = tmax_ecu,
prcp = prcp_ecu,
output_dir = output_dir_bioclim,
overwrite = TRUE
)
# Plot bio01 y bio12
plot(bioclim_ecu[[c("bio01", "bio12")]])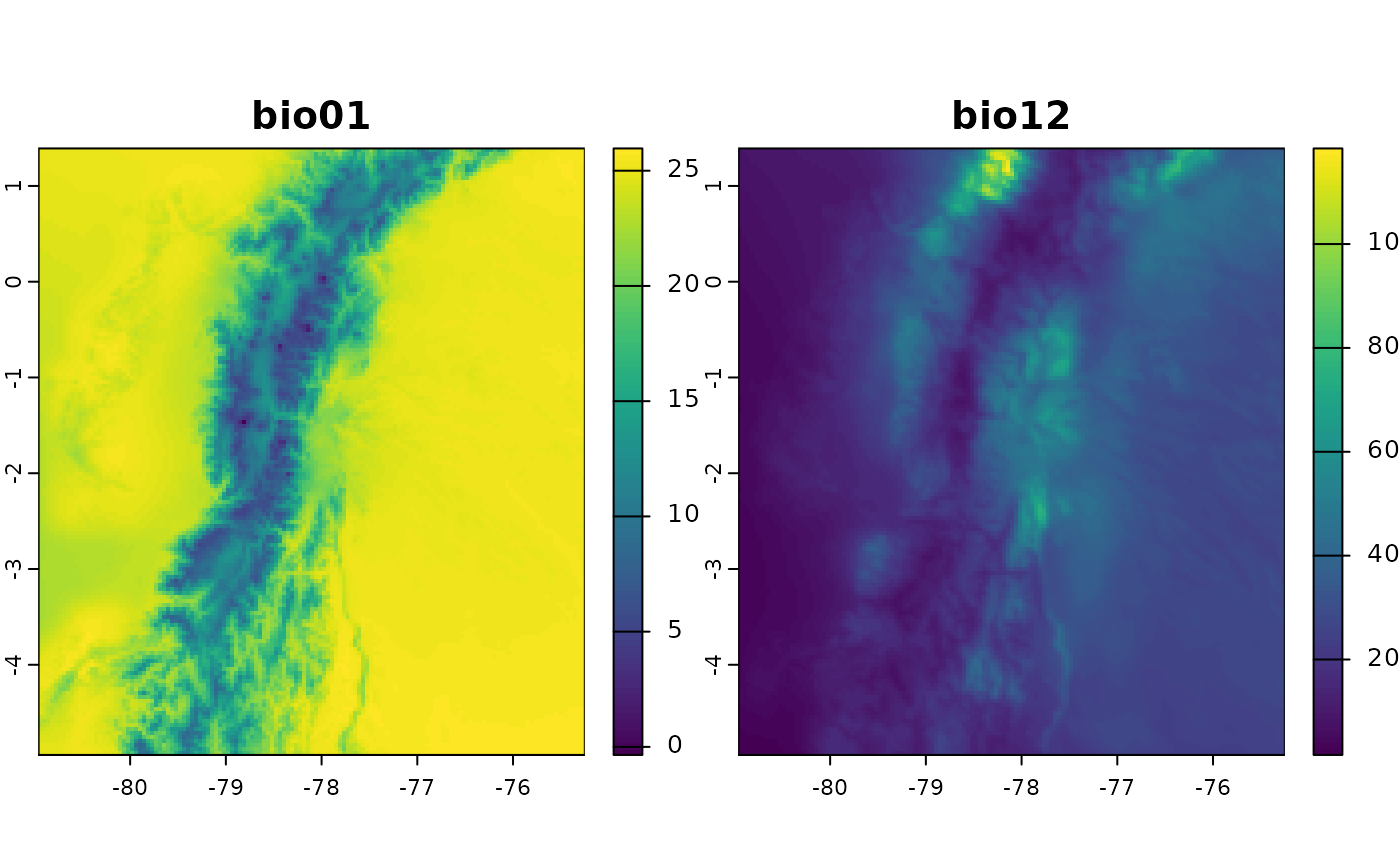
Usando temperatura promedio como input
El paquete fastbioclim también ofrece la opción de usar
temperatura promedio (definida con el parámetro tavg) para
el cálculo de variables bioclimáticas.
# Temperatura promedio
tavg_ecu <- system.file("extdata/ecuador/", package = "egdata.fastbioclim") |>
list.files("tavg", full.names = TRUE) |> rast()
# Definir directorio donde se guardarán los rasters
output_dir_bioclim_v2 <- file.path(tempdir(), "bioclim_ecuador_v2")
bioclim_ecu_v2 <- derive_bioclim(
bios = 1:19,
tavg = tavg_ecu,
tmin = tmin_ecu,
tmax = tmax_ecu,
prcp = prcp_ecu,
output_dir = output_dir_bioclim_v2,
overwrite = TRUE
)
# Diferencia entre bio01s cuando se usa tavg
plot(bioclim_ecu_v2[["bio01"]] - bioclim_ecu[["bio01"]])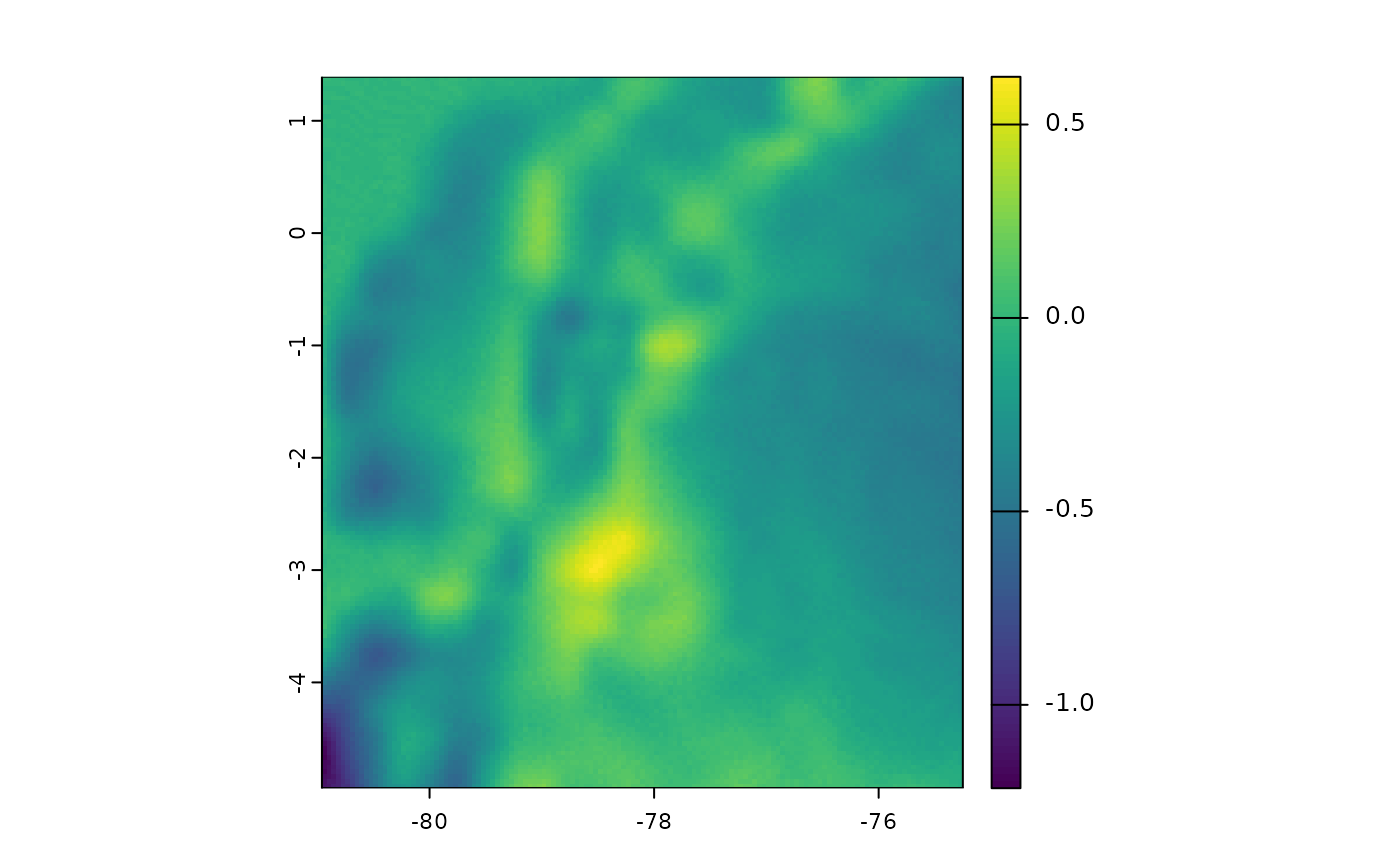
Seleccionar un grupo de variables
Muchas veces no es necesario utilizar todas las variables
bioclimáticas en nuestros análisis. Por este motivo, y a diferencia de
biovars(), se puede definir en el parámetro
bios el número que identifica cada una de las variables
bioclimáticas. De esta forma no es necesario obtener todas las 19
variables para luego solo seleccionar las variables de interés. En el
siguiente ejemplo solo se obtendrán cuatro variables (bio05, bio06,
bio13 y bio14). Este ejemplo es algo más rápido, ya que no es necesario
calcular internamente los trimestres más cálidos/fríos o
secos/húmedos.
bios4_ecu <- derive_bioclim(
tmin = tmin_ecu,
tmax = tmax_ecu,
prcp = prcp_ecu,
bios = c(5, 6, 13, 14),
overwrite = TRUE
)
plot(bios4_ecu)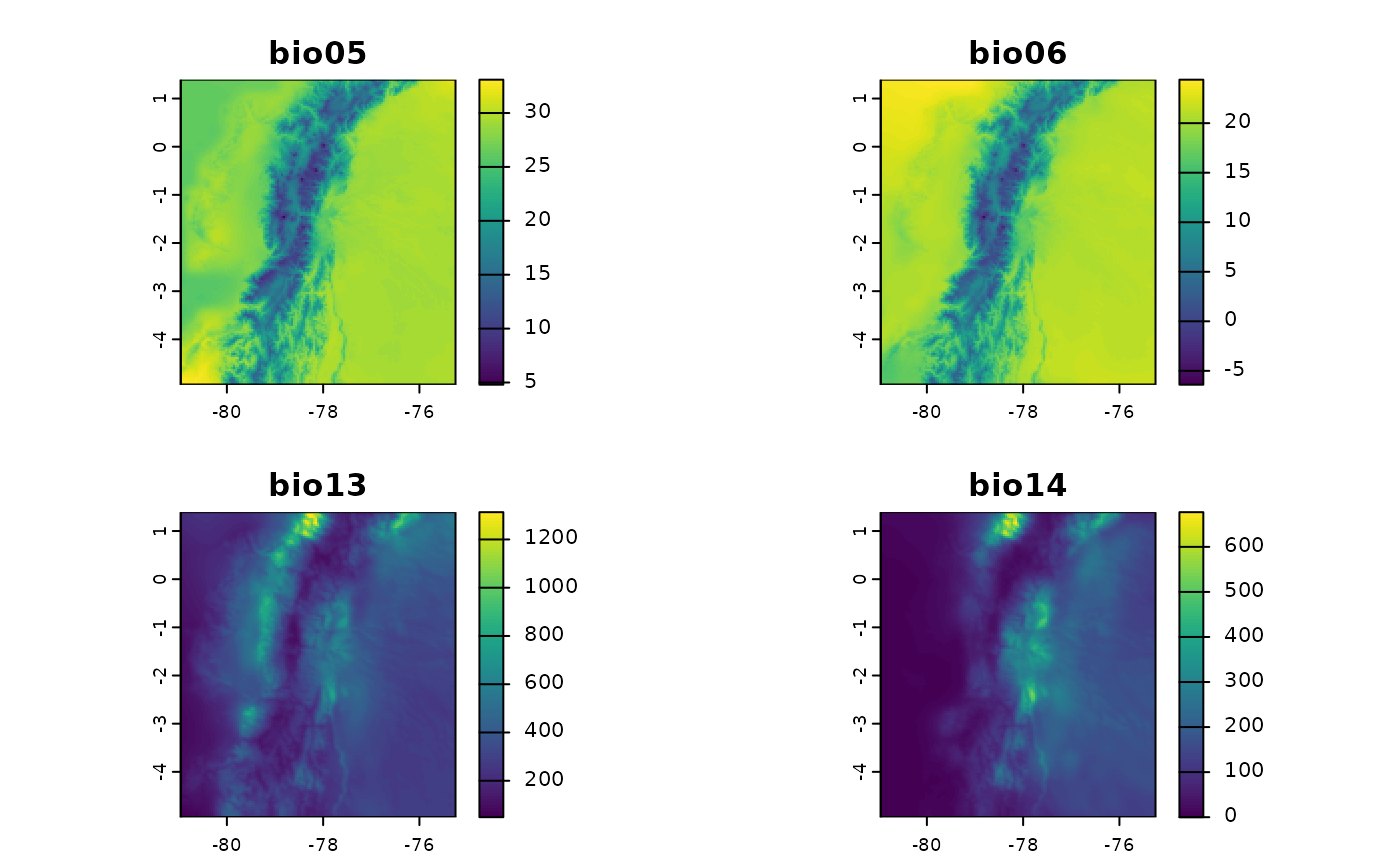
Construir resúmenes con otras variables
Otra funcionalidad importante de fastbioclim es la
opción de obtener estadísticas similares a las bioclimáticas pero con
otras variables. Como ejemplo, realizaremos resúmenes de variables
promedio mensuales de viento. Para las variables interactivas
trimestrales, usaremos los trimestres más húmedos y secos.
wind_ecu <- system.file("extdata/ecuador/", package = "egdata.fastbioclim") |>
list.files("wind", full.names = TRUE) |> rast()
wind_dir_ecu <- file.path(tempdir(), "wind_ecuador")
ecu_stats <- derive_statistics(
variable = wind_ecu,
stats = c("mean", "max", "min", "stdev", "max_period", "min_period"),
inter_variable = prcp_ecu,
inter_stats = c("max_inter", "min_inter"),
prefix_variable = "wind",
suffix_inter_max = "wettest",
suffix_inter_min = "driest",
overwrite = TRUE,
output_dir = wind_dir_ecu
)
plot(ecu_stats)
También se pueden construir subconjuntos de variables, en este caso vamos a construir variables de viento, pero solo basado en la interacción con la temperatura, las cuales corresponden a “Viento en el trimestre más cálido” y “Viento en el trimestre más frío”.
ecu_stats_v2 <- derive_statistics(
variable = wind_ecu,
stats = NULL,
inter_variable = tavg_ecu,
inter_stats = c("max_inter", "min_inter"),
prefix_variable = "wind",
suffix_inter_max = "warmest",
suffix_inter_min = "coldest",
overwrite = TRUE,
output_dir = wind_dir_ecu
)
plot(ecu_stats_v2)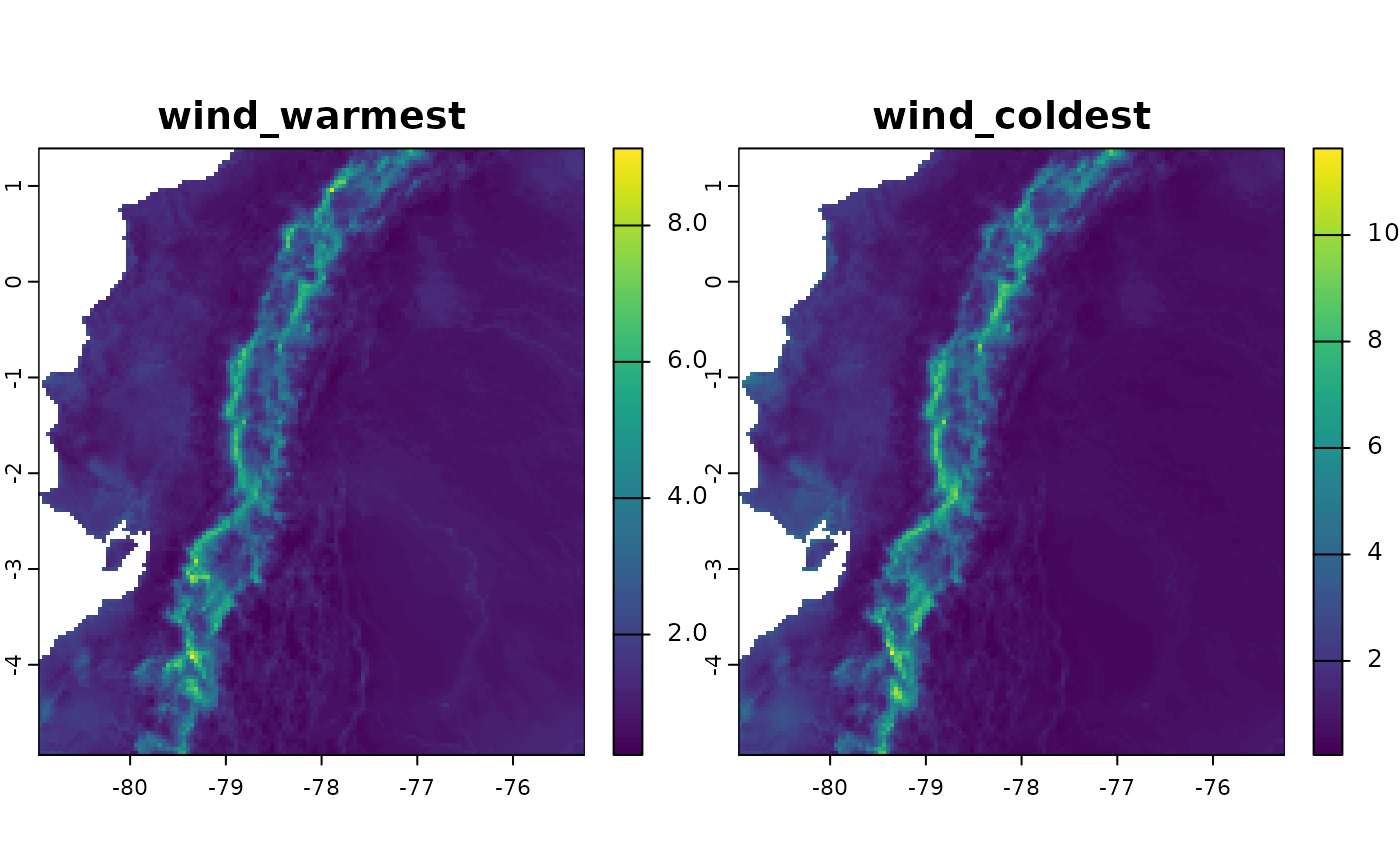
Construir Neotrópico: 35 variables
Basado en la nomenclatura de ANUCLIM 6.1 (Xu & Hutchinson, 2012),
derive_bioclim() también ofrece la opción de crear
variables bioclimáticas basadas en índices de humedad (moisture
en inglés) y radiación solar. En este caso, vamos a construir las 35
variables bioclimáticas para la extensión que cubra el Neotrópico.
En este caso, el método “auto” debería usar la creación de las
variables usando el método de “tiles” (mosaicos). Pero también se puede
forzar a usar dicho método usando el parámetro
method="tiled". Este método dividirá el área de interés en
mosaicos usando los grados decimales definidos en el parámetro
tile_degrees (5 es el valor por defecto).
Paralelización: También es importante mencionar que
el método ‘tiled’ se puede paralelizar usando
future::plan(). Para más información consulte la
documentación de dicho paquete.
Barra de progreso: La barra de progreso está
disponible usando el paquete progressr. Para activarla es
necesario usar la función progressr::handlers() o
progressr::with_progress(). Para más información consulte
la documentación de dicho paquete.
# Obtener lista de rasters y crear un spatRaster para cada variable
# Temperatura promedio
tavg_neo <- system.file("extdata/neotropics/", package = "egdata.fastbioclim") |>
list.files("tavg", full.names = TRUE) |> rast()
# Temperatura mínima
tmin_neo <- system.file("extdata/neotropics/", package = "egdata.fastbioclim") |>
list.files("tmin", full.names = TRUE) |> rast()
# Temperatura máxima
tmax_neo <- system.file("extdata/neotropics/", package = "egdata.fastbioclim") |>
list.files("tmax", full.names = TRUE) |> rast()
# Precipitación
prcp_neo <- system.file("extdata/neotropics/", package = "egdata.fastbioclim") |>
list.files("prcp", full.names = TRUE) |> rast()
# Radiación solar
srad_neo <- system.file("extdata/neotropics/", package = "egdata.fastbioclim") |>
list.files("srad", full.names = TRUE) |> rast()
# Índice climático de humedad
mois_neo <- system.file("extdata/neotropics/", package = "egdata.fastbioclim") |>
list.files("cmi", full.names = TRUE) |> rast()
# Definir directorio donde se guardarán los rasters
output_dir_neo <- file.path(tempdir(), "bioclim_neotropics")
# Activar barra de progreso
# progressr::handlers(global = TRUE)
# Definir plan de paralelización
# future::plan("multisession", workers = 4)
# Obtener las 35 variables para el Neotrópico
bioclim_neo <- derive_bioclim(
bios = 1:35,
tavg = tavg_neo,
tmin = tmin_neo,
tmax = tmax_neo,
prcp = prcp_neo,
srad = srad_neo,
mois = mois_neo,
method = "tiled",
tile_degrees = 20,
output_dir = output_dir_neo,
overwrite = TRUE
)
print(bioclim_neo)## class : SpatRaster
## size : 2120, 1978, 35 (nrow, ncol, nlyr)
## resolution : 0.04166667, 0.04166667 (x, y)
## extent : -117.1251, -34.70847, -55.60847, 32.72486 (xmin, xmax, ymin, ymax)
## coord. ref. : lon/lat WGS 84 (EPSG:4326)
## sources : bio01.tif
## bio02.tif
## bio03.tif
## ... and 32 more sources
## names : bio01, bio02, bio03, bio04, bio05, bio06, ...
## min values : -15.08724, 0.06966146, 1.224817, 5.000592, -6.597656, -26.50000, ...
## max values : 30.27604, 18.90247536, 91.692070, 836.565613, 42.875000, 26.29688, ...Ejemplo con región definida por el usuario
Otro parámetro útil en el paquete fastbioclim es la
opción de proveer un objeto ‘sf’ para delimitar y enmascarar un área de
interés. El cálculo de las variables bioclimáticas solo se realizará en
dicha área.
# Obtener áreas de interés
mex <- qs2::qs_read(system.file("extdata/mex.qs2", package = "egdata.fastbioclim"))
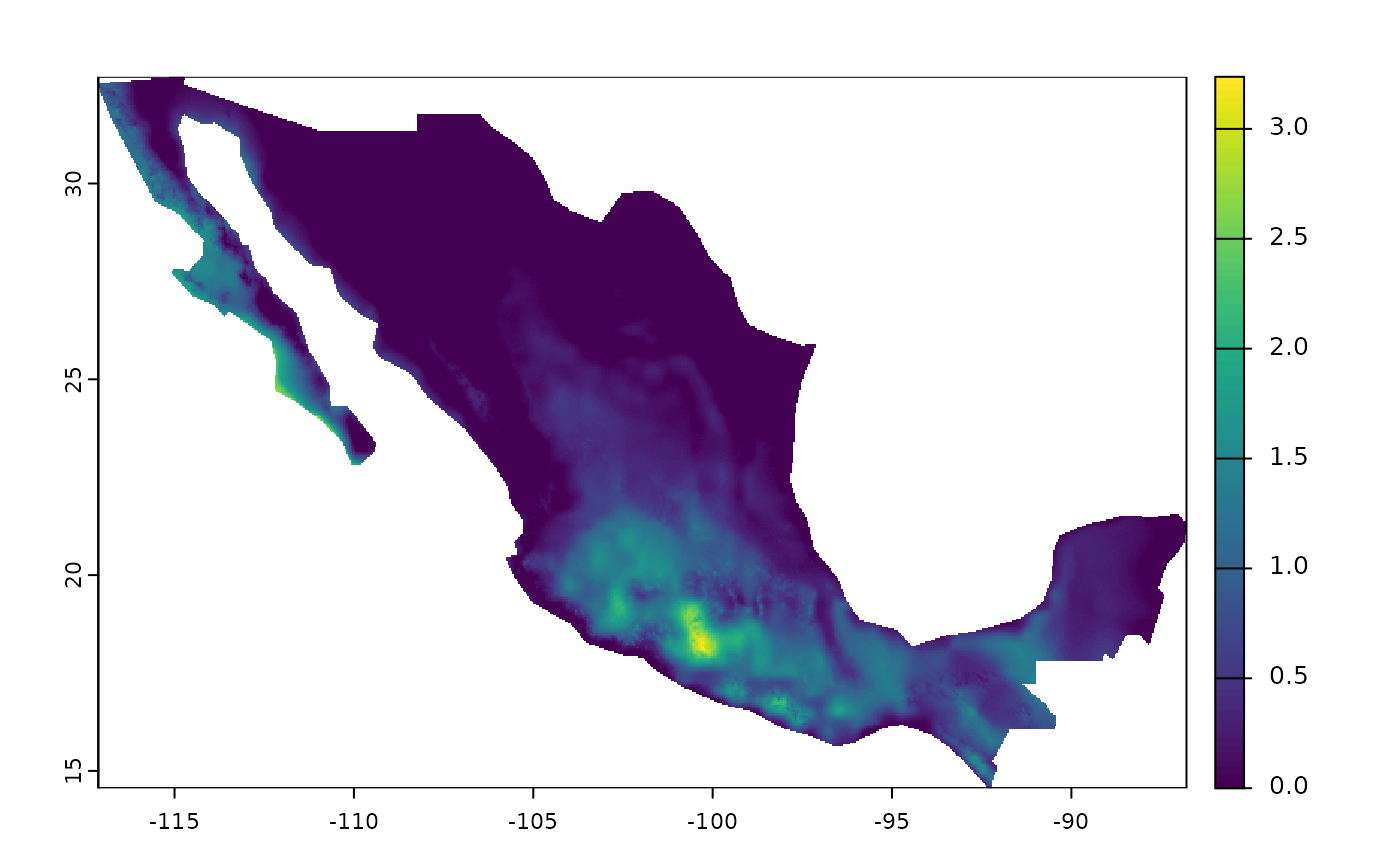
# Obtener solo bio10
bio10_mex <- derive_bioclim(
bios = 10,
tmax = tmax_neo,
tmin = tmin_neo,
user_region = mex,
overwrite = TRUE,
output_dir = file.path(tempdir(), "bio10_mex")
)
plot(bio10_mex)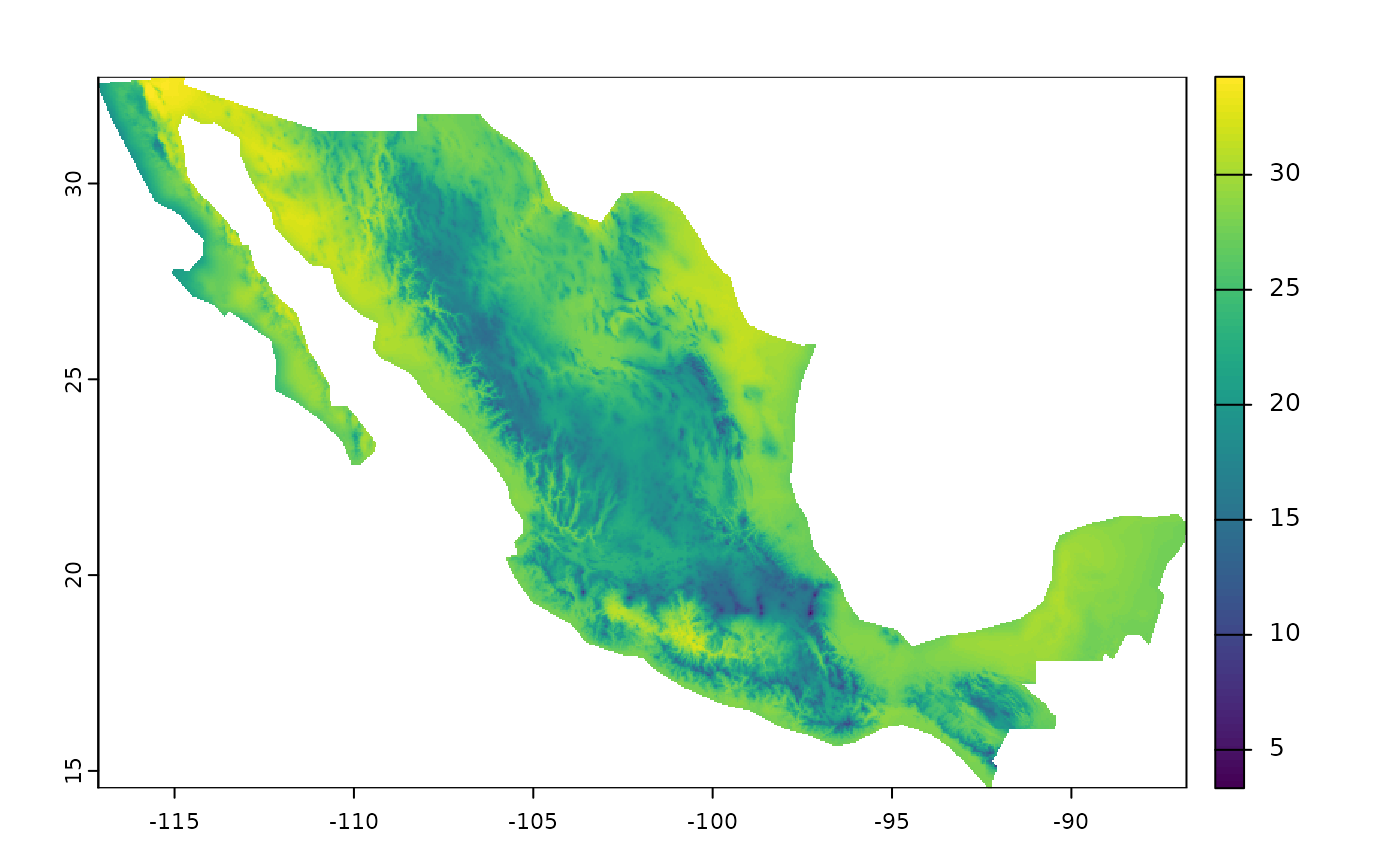
Ejemplo con variable estática
Una opción avanzada dentro del paquete es la posibilidad de determinar variables estáticas para las variables máximas y mínimas por meses (o unidades) y períodos. Esto puede llegar a ser útil si las preguntas de investigación están relacionadas con un tiempo específico (p.ej. estaciones del año) o en la construcción de series de tiempo.
En este caso, vamos a crear nuevamente la variable bio10 en México,
pero usando como referencia el trimestre de junio, julio y agosto. Para
ello, debemos crear un spatRaster que defina el período de interés. En
fastbioclim siempre se referencia el primer mes del período
o la unidad. En este caso, dicho mes corresponde al número 6. Por lo
cual, crearemos un raster lleno de este número, para luego ser usado
como referencia en la creación de la variable ‘bio10’.
# Crear un raster de 6s para el neotrópico
warmest <- tavg_neo[[1]]
warmest[!is.na(warmest)] <- 6
names(warmest) <- "warmest_period"
# Importante: Para el método 'tiled' los raster deben estar guardados en disco
terra::writeRaster(warmest, file.path(tempdir(), "warmest_static.tif"), overwrite = TRUE)
warmest <- terra::rast(file.path(tempdir(), "warmest_static.tif"))
plot(warmest)
# Obtener solo bio10
bio10_war <- derive_bioclim(
bios = 10,
tmax = tmax_neo,
tmin = tmin_neo,
user_region = mex,
warmest_period = warmest,
overwrite = TRUE,
output_dir = file.path(tempdir(), "bio10_war")
)
# Diferencias en bio10 de México
plot(bio10_mex - bio10_war)